3.1 ORGANISMOS PROCARIÓTICOS
Los procariontes o procariotas (del inglés «procaryote» y
éste de las raíces griegas «pro», inferior, y «karyon», núcleo; es decir,
«animal inferior, sin núcleo») son células sin núcleo diferenciado (es decir,
cuyo ADN está en el citoplasma) y los organismos constituidos por ellas. Las
células con núcleo diferenciado rodeado de membrana nuclear se llaman
eucariotas; esta distinción entre eucariontes y procariontes se considera la de
mayor importancia en el reino animal. Las células procariotas fueron los únicos
seres vivos en la Tierra durante 2.000 millones de años hasta la aparición de
las eucariotas.
Los organismos procariontes son en su mayoría
unicelulares, bacterias, hasta el punto de que ambos términos se han usado a
veces como sinónimos. Carl Woese propuso clasificarlos en Bacteria y Archaea
(originalmente Eubacteria y Archaebacteria) creyendo que ambos tienen orígenes
distintos. Esta controvertida clasificación, finalmente aceptada por la
comunidad científica, se denomina «sistema de los tres dominios.
Las células procariontes carecen de citoesqueleto y
compartimentos celulares con membrana como vacuolas, retículo endoplásmico,
mitocondria y cloroplastos. Tienen un cromosoma pero pueden además presentar
pequeñas piezas circulares de ADN denominadas plásmidos. La reproducción es
exclusivamente asexual mediante fisión binaria.
Hay 2 tipos de organización celular: uno, elemental más
primitivo, recibe el nombre de célula procariota o protozito. Corresponde a la
estructura de la célula bacteriana y de los cianofitos (que actualmente se
consideran bacterias). Los seres vivos integrados por células procariotas
constituyen actualmente el reino de los monerados (o también protistas
inferiores). El resto de los seres vivos, ya sean unicelulares (protistas) o
pluricelulares (vegetales o metafitas y animales o metazuarios), están
constituidos por una o muchas células eucariotas o eucitos, tienen núcleo
normal
En general, los genomas bacterianos tienen un tamaño
inferior a 5 Mb, aunque en el caso de Bacillus megaterium el genoma tiene un
tamaño de 30 Mb. La mayor parte del genoma corresponde a genes.
3.1.1 ADN CIRCULAR
Adn Circular
Cualquiera de las
moléculas de ADN covalentemente cerradas que se encuentran en bacterias, muchos
virus, mitocondrias, plástidos, y plásmidos. También se han observado ADNs
pequeños, circulares y polidispersos en un número de organismos eucarióticos y
se ha sugerido que tienen homología con el ADN cromosómico y en la capacidad de
insertarse en él, y escindirse del ADN cromosómico. Es un fragmento de ADN
formado por un proceso de formación y de deleción de un asa , que contiene una
región constante de la cadena pesada mu y la parte 3 de la región de cambio mu.
El adn circular es un producto normal de la reordenación entre los segmentos
del gen que codifica las regiones variables de las cadenas ligeras y pesadas de
las inmunoglobulinas, así como de los receptores de las células T.
GENOMAS PROCARIÓTICOS;
Cairns (1963) interpretó que el cromosoma de E. coli era
circular, que se replicada de modo semiconservativo, que existía un punto de
inicio de la replicación, un origen, y un punto de crecimiento (PC). Sin
embargo, esta interpretación fue errónea, ya que en E. coli existe un solo
punto de iniciación de la Replicación (Ori C) pero existen dos puntos de
crecimiento (PC), ya que la replicación en E. coli, como veremos más adelante
es bidireccional.
El genoma de la mayoría de los procariontes está formado
por un único cromosoma. Normalmente es una molécula de DNA de doble cadena
cerrada y circular.
Los genes bacterianos estan muy agrupados, con distancias
intergénicas muy pequeñas, y los intrones son muy escasos.
3.1.2 PROTEÍNAS
ASOCIADAS
El DNA en bacterias se
organiza en un nucleoide, que se forma por un superenrrollamiento.
Arquitectura del genoma en procariotas
En E.coli existen una serie de proteinas relacionadas con
el empaquetamiento del DNA que reciben el nombre de proteinas similares a
histonas p.e. histone-like nucleoid structuring protein (H-NS), integration
host factor (IHF) etc.
su similitud con las histonas de los eucariotas es muy
baja (a excepción de las proteinas HU).
Estas proteinas posiblemente son capaces de remodelar el
grado de compactación del DNA del nucleoide, influyendo sobre la expresión
génica.
3.1.3 ADN EXTRACROMOSÓMICO
En diversos experimentos ha quedado demostrado que la
transmisión de la información genética es a través de genes y cromosomas
nucleares. Sin embargo investigaciones esporádicas indicaron que algunos
elementos extranucleares o citoplásmicos podían actuar como agentes de
trasmisión hereditaria.
En algunos organelos extranucleares como por ejemplo en
las mitocondrias y como también en los simbiontes extranucleares se demostró la
existencia y el funcionamiento del ADN citoplásmico como material transportador
de información genética.
Asimismo, se registraron algunos posibles casos de
herencia independiente del ADN y ciertas influencias del citoplasma del óvulo
que modifica el fenotipo de la descendencia.
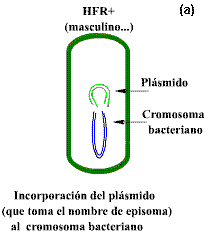
ADN cromosómico: cromosoma bacteriano
ADN extra cromosómico: elemento facultativo (plásmidos,
Bacteriófagos atemperados, etc.)
Libres o unidos al cromosoma (episomas)
Propiedades importantes pero no esenciales para la vida
Bacteriana
Replicación independiente del núcleo
Transferencia a otras células o herencia a células
hijas
Contienen información para su replicación y proteínas
Reguladoras.
Adquisición por conjugación, transducción y posibilidad
de perderlos (curación)
3.1.3.1 PLÁSMIDOS
Genomas plásmidicos
Los plásmidos son moléculas circulares de ADN que se
replican de manera independiente al cromosoma de la célula hospedera. De manera
natural se encuentran en las bacterias en tamaños que van desde 5,000 hasta
400,000 pb.
Pueden ser introducidos en las células bacterianas por un
proceso denominado transformación. Las células y el plasmido se incubas juntos
a 0°C en soluciones de cloruro de calcio, posteriormente se da un incremento de
temperatura al medio de entre 37 y 43 °C, alternativamente se puede utilizar un
choque de corriente eléctrica en una técnica denominada electroporación.
Los plásmidos son moléculas de ADN extracromosómico
circular o lineal que se replican y transcriben independientes del ADN cromosómico.
Están presentes normalmente en bacterias, y en algunas ocasiones en organismos
eucariotas como las levaduras. Su tamaño varía desde 1 a 250 kb. El número de
plásmidos puede variar, dependiendo de su tipo, desde una sola copia hasta
algunos cientos por célula. El término plásmido fue presentado por primera vez
por el biólogo molecular norteamericano Joshua Lederberg en 1952
Las células bacterianas contienen comúnmente pequeños
elementos de DNA que no son esenciales para las operaciones básicas de la
célula. Estos componentes se denominan plasmidos.
Los plasmidos no pueden vivir fuera de la célula.
Los plasmidos bacterianos contienen a menudo genes que
son útiles para la célula huésped, p.e. confieren resistencia a antibióticos, promueven la fusión o producen
toxinas.
los plasmidos bacterianos suelen ser circulares, aunque
se han encontrado algunos lineales.
Existencia autónoma del cromosoma bacteriano (raramente
se insertan).
Portadores de genes no presentes en el cromosoma
bacteriano p.e. genes de resistencia a antibióticos.
Un mismo plásmido puede hallarse en especies distintas de
bacterias.
3.1.3.2 BACTERIÓFAGOS
La historia del descubrimiento de los bacteriofagos ha
sido objeto de muchos debates, los cuales incluyen una controversia sobre quién
fue su descubridor. En 1913, el bacteriólogo británico Frederick Twort
descubrió un agente bacteriolítico que infectaba y mataba a las bacterias,
pero, no fue capaz de saber qué era exactamente dicho agente, señalando en una
de sus hipótesis que, entre otras posibilidades, podría tratarse de un virus.
No fue sino hasta 1917, cuando el microbiólogo canadiense Félix d'Herelle
anunció el descubrimiento de "un invisible antagonista microbiano del
bacilo de la disentería", al cual afirmó que se trataba de un virus al
cual llamó bacteriófago. Los trabajos de d'Herelle iniciaron desde 1910 y en
contraste con Twort y algunos otros científicos que habían reportado fenómenos
similares, tuvo pocas dudas sobre la naturaleza del fenómeno que estaba
observando y afirmó que se trataba de un virus que parasitaba a la bacteria. El
nombre bacteriófago lo formó de la palabra "bacteria" y "phagein"
(comer o devorar, en griego), implicando que los fagos "comen" o
"devoran" a las bacterias.
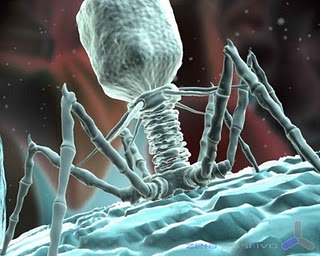
Los bacteriófagos (también llamados fagos -del griego
φαγητόν (phagētón), «alimento, ingestión») son virus que infectan
exclusivamente a bacterias.
Al igual que los virus que infectan células eucariotas,
los fagos están constituidos por una cubierta proteica o cápside en cuyo
interior está contenido su material genético, que puede ser ADN o ARN de simple
o doble cadena, circular o lineal (en el 95% de los fagos conocidos es ADN de
doble cadena), de 5.000 a 500.000 pares de bases. El tamaño de los fagos oscila
entre 20 y 200 nm aproximadamente.
Los fagos pueden generar el ciclo lítico o el ciclo
lisogénico, aunque muy pocos son capaces de llevar a cabo ambos.
En el ciclo lítico, las células hospedadoras del fago son
lisadas (destruidas) tras la replicación y encapsulación de las partículas
virales, de forma que los nuevos virus quedan libres para llevar a cabo una
nueva infección.
Por el contrario, en el ciclo lisogénico no se produce la
lisis inmediata de la célula. El genoma del fago puede integrase en el ADN
cromosómico de la bacteria hospedadora, replicándose a la vez que lo hace la
bacteria o bien puede mantenerse estable en forma de plásmido, replicándose de
forma independiente a la replicación bacteriana.
En cualquier caso, el genoma del fago se transmitirá a
toda la progenie de la bacteria originalmente infectada. El fago queda así en
estado de latencia hasta que las condiciones del medio se vean deterioradas:
disminución de nutrientes, aumento de agentes mutagénicos, etc. En este
momento, los fagos endógenos o profagos se activan y dan lugar al ciclo lítico
que termina con la lisis celular.
3.1.3.3 TRANSPOSONES
Transposición:
Transposon. Secuencia de DNA que puede moverse de un
lugar a otro del cromosoma, insertar copias adicionales de ella misma en otros
puntos o pasar de un cromosoma a otro. Tramo de DNA que puede incorporarse en
otras moléculas de DNA en lugares donde no hay homología de secuencia. Tienen
un tamaño entre 1 y 40 kb. Codifican todas las enzimas necesarias para su
inserción. Pueden desplazarse dentro de un cromosoma o entre cromosomas.
Elemento genético móvil que puede moverse de una
localización genómica a otra, gracias a la presencia de secuencias repetidas
cortas que lo flanquean y que es capaz de replicar e insertar una copia en un
lugar nuevo en el genoma.
Un transposón o elemento genético transponible es una
secuencia de ADN que puede moverse de manera autosuficiente a diferentes partes
del genoma de una célula, un fenómeno conocido como transposición. En este
proceso, se pueden causar mutaciones y cambio en la cantidad de ADN del genoma.
Anteriormente fueron conocidos como "genes saltarines" y son ejemplos
de elementos genéticos móviles
La transposición es el fenómeno por el cual un elemento de
DNA cambia de ubicación física en el cromosoma sin aprovechar ningún tipo de
homología de secuencia, sino a través de una proteína específica denominada
transposasa. Las características que distinguen la transposición de la
recombinación son:
No requiere homología de DNA entre la secuencia dadora y
la aceptora, aunque hay puntos calientes en regiones ricas en AT
Se produce una duplicación de la secuencia aceptora
(entre 3 y 12 pb) antes y detrás del transposón
la intervención de la transposasa y, en algunos casos, la
resolvasa.
La transposición puede reestructurar drásticamente la
organización del cromosoma hospedador. Además, puede alterar la expresión de un
gen, bien inactivándolo, bien sobreexpresándolo.
Su presencia la demostró Barbara McClintok en los años
50.
Transposones procarióticos
- Tipos
La clasificación se basa principalmente en los genes que
aparecen
Tipo I
Se conocen como transposones simples o secuencias de
inserción (IS) lo que en 700 o 2000 pb contienen el gen TnpA que codifica la
transposasa flanqueado por dos secuencias invertidas repetidas cortas (15 a 25
pb).
Tipo II
Contienen al menos tres genes: una transposasa (TnpA),
una resolvasa (TnpR) y un gen que suele ser de resistencia a antibióticos. Eso
se encuentra flanqueado por secuencias repetidas invertidas (IR) a la izquierda
(IR-L) y a la derecha (IR-R).
Tipo III
Aquellos fagos que, en lugar de insertarse en el genoma
por recombinación —lo normal—, lo hace mediante transposición. El más conocido
es el fago μ.
La gran mayoría de los transposones eucarióticos utilizan
RNA como intermediario de la transcripción.
3.2
ORGANISMOS EUCARIÓTICOS
Genomas nucleares eucarióticos
En los organismos eucarióticos, la mayoría de los genes
se encuentra en los cromosomas del núcleo. Las especies eucarióticas se
clasifican como diploides (dos series de cromosomas en el núcleo), o son
haploides (1 sola serie de cromosomas en el nucleo), la mayoría de algas y
hongos son haploides y el resto de eucariotas (incluyendo plantas y animales)
son diploides.
3.2.1 ADN
LINEAL Y EMPAQUETAMIENTO
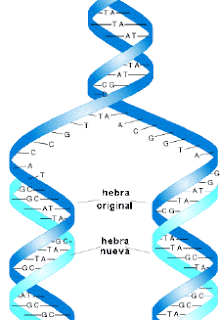
El ADN está formado por pequeñas piezas llamadas
nucleótidos, éstos a su vez están formados por una base nitrogenada (purica o
pirimidinica), un azúcar (en el caso del ADN es la desoxirribosa, la primera
letra de esta azúcar está incluida en la palabra ADN) y un grupo fosfato. Cada
uno de estos nucleótidos se unen entre sí formando una cadena, ésta puede ser
muy larga (más de un metro). Esta es una cadena única de ADN, o como tu dices
ADN lineal. Biológicamente ésta no puede funcionar por sí sola, necesita una
cadena complementaria (otra cadena lineal) que se unirá a la primera por
puentes de hidrógeno. Así que hasta el momento tenemos dos cadenas unidas,
semejarían una escalera. Ahora, esta doble cadena de ADN no puede estar de
forma recta, imagina que tienes una escalera y eres tan fuerte que puedes
doblarla desde un extremo y la haces de forma helicoidal, estonces quedaría
como una escalera de caracol. Pues al ADN le sucede lo mismo, giran las cadenas
formando una doble hélice en forma helicoidal, pero ahí no termina todo.

Una célula humana contiene alrededor de 2 metros de DNA
(1 metro por cada serie cromosomica). El cuerpo humano está constituido por
unas 1013 células y cada célula es diploide, por lo que contiene en total unos
2x1013 metros de DNA.
3.2.1.1 HISTONAS
Las proteínas responsables del empaquetamiento del ADN se
denominan histonas. Estas proteínas
forman parte del alrededor de la mitad de la masa de la cromatina. Hay 5 clases
principales de histonas: H1, H2A, H2B, H3 y H4,
todas ellas contienen una gran cantidad de residuos cargados
positivamente ( Arg y Lys).
el empaquetamiento ocurre en el núcleo donde el DNA se
condensa en 46 cromosomas.
La mezcla completa de materiales de los que se compone el cromosoma se conoce
como cromatina. Se trata de DNA y proteínas.
Las histonas son proteínas asociadas al DNA en los
nucleososmas. Los nucleosomas (10 nm) están formados por un octamero compuesto
por dos unidades de cada una de las histonas (H2A, H2B, H3 Y H4).
3.2.1.2 SOLENOIDES
El DNA (asociado a las histonas) da dos vueltas alrededor
de cada octamero de los nucleosomas, el nucleosoma es una forma distendida, de
una forma muy enrollada denominado solenoide (30 nm) , el solenoide mantiene su
forma mediante otra histona H1.
Para conseguir el primer nivel de empaquetamiento el DNA
se enrolla alrededor de las histonas, que actúan, como bobinas de un hilo, un
nuevo enrollamiento genera la conformación del solenoides.
3.2.1.3
CROMOSOMAS
Enrollamientos de orden superior.
Se denomina cromosoma (del griego χρώμα, -τος chroma,
color y σώμα, -τος soma, cuerpo o elemento) a cada uno de los pequeños cuerpos
en forma de bastoncillos en que se organiza la cromatina del núcleo celular
durante las divisiones celulares (mitosis y meiosis). En las células eucariotas
y en las arqueobacterias (a diferencia que en las células procariotas), el ADN
siempre se encontrará en forma de cromatina, es decir asociado fuertemente a
unas proteínas denominadas histonas
Los cromosomas se encuentran muy enrollados, si un
solenoide tiene de diámetro 30 nm, un cromosoma condensado tiene 700 nm. Para
esto el solenoide se enrolla sobre un esqueleto proteico compuesto de la enzima
topoisomerasa II, que es capaz de pasar una cadena de DNA a través de otra.
COMO SE EMPAQUETA EL ADN.
1) el DNA se enrolla sobre los nucleosomas que actúan
como bobinas de hilo.
2) los nucleosomas se enrollan formando un solenoide.
3) el solenoide forma lazos anclados a un esqueleto
central.
4) el esqueleto unido a los lazos se dispone como un
solenoide gigante.
3.2.2
COMPLEJIDAD DEL GENOMA
un gen es una región de DNA cromosómico que puede
trascribirse en una molécula de RNA funcional en el momento y lugar adecuados
del proceso de desarrollo de un organismo.
El hecho de definir con precisión que es un gen puede
dificultarse ya que muchos genes eucarioticos contienen segmentos de DNA,
llamados intrones, que se encuentran intercalados en la región transcrita del
gen. Los intrones no contienen información para al formación del producto
génico correspondiente. (p.e proteína). Se transcriben junto con las regiones
codificantes (llamadas exones) pero luego son eliminados del transcrito
inicial.
La correcta secuencia de nucleótidos de los intrones, de
la región reguladora y de la región codificante es necesaria para crear un
trascrito del tamaño adecuado, en el momento y lugar adecuado y estas tres
partes deben considerarse como una unidad funcional completa, en otras palabras
como parte de un gen.
Los análisis de secuencia han demostrado que hay DNA
entre los genes, de función desconocida la mayor parte. El tamaño y la
naturaleza de este DNA dependen del genoma. En hongos, este DNA intergénico es
pequeño, pero en mamíferos es muy grande.
Los tamaños de los genomas se miden en unidades formadas
por miles de bases de nucleotidos (llamados kilobase, kb) o millones de pares
de nucleotidos (megabases, mb),
3.2.3
ADN MITOCONDRIAL
Los cromosomas de mitocondrias y cloroplastos son de DNA
de doble cadena.
Las mitocondrias
se encuentran en todos los seres eucariotas aerobios; contienen las
enzimas para la mayor parte de las reacciones oxidativas que generan energia
para las funciones celulares.
Actualmente se conocen las secuencias completas del ADN
de varios genomas mitocondriales. Al ADN mitocondrial se lo conoce como ADNmt
(mtDNA).
La estructura del genoma mitocondrial es circular como es
el del genoma bacteriano. Se trata de una mol. circular de ADN, helicoidal, con
doble hebra, y supercondensada. Se conocen algunos pocos casos de genomas
mitocondriales de forma lineal. En muchos casos, el contenido de GC
(guanina-citosina) del ADNmt difiere en gran medida del ADN nuclear, y esto
permite separar el ADNmt del nuclear por centrifugacion en un gradiente de cloruro de cesio.
En los animales, el genoma mitocondrial generalmente es
menor a 20 kb (kilobases); por ej. en el hombre el ADNmt tiene 16.569 bp (pares
de bases). Por su parte, el ADNmt de una levadura contiene cerca de 80.000 bp
(80 kb), mientras que en las plantas varia entre 100 y 2.000 kb.
3.3
ORGANIZACIÓN GENÓMICA VIRAL
Los genomas virales son muy distintos entre si, muchos
estan compuestos de ADN, que cuando estan empaquetados puede ser de cadena
sencilla y cadena doble. Algunos virus como el HIV (retrovirus) contiene
genomas de RNA, algunos de cadena sencila y otros de cadena doble. Algunos
genomas virales contienen DNA y RNA circulares.
Independiente del genoma del virus hay siempre una fase
intracelular del ciclo infectivo, en la cual este genoma se convierte en DNA de
cadena doble.
Los virus pueden tener DNA duplex, DNA monocatenario, RNA
monocatenario o RNA duplex.
El tamaño de los virus está comprendido entre 20 y 300
nm. Ya que la mayoría miden menos de 250 nm, límite de resolución del
microscopio óptico, sólo son visibles con ayuda del microscopio electrónico.
Los virus están compuestos de un núcleo central formado
por ácido nucleico (DNA o RNA, pero nunca los dos en el mismo virión) rodeado
por una proteína que constituye la cápsida. El núcleo central y la cápsida
forman conjuntamente la nucleocápsida del virión.
BIBLIOGRAFIA
* antología
biología molecular 2012

.png)














.jpg)
